Tijdens het uitvoeren van genetische studies komen we vaak onvoldoende RNA-monsters tegen, bijvoorbeeld voor het bestuderen van kleine anatomische orale tumoren, zelfs eencellige monsters en monsters van specifieke genmutaties die op zeer lage niveaus in menselijke cellen worden getranscribeerd.Natuurlijk, voor de COVID-19-test, als de wattenstaafjes niet op de juiste plaats zijn of niet vaak genoeg tijdens de bemonstering, zal de steekproefomvang erg klein zijn. Daarom kwam de Commissie voor Gezondheid en Gezinsplanning twee dagen geleden naar buiten en geslaagd voor de test en als de nucleïnezuurmonsternemer geen zes monsters heeft genomen, kunt u dit melden.
De gevoeligheid van het reagens is belangrijk omdat we dit of dat probleem hebben, dus wat kunnen we doen om de gevoeligheid van RT-PCR te verbeteren?
Laten we, voordat we mogelijke oplossingen bespreken, twee grote complicaties noemen met de situatie die we zojuist noemden.
Allereerst maken we ons zorgen over RNA-verlies als we slechts een paar celpopulaties in onze steekproef hebben.Als traditionele scheidings- en reinigingsmethoden worden gebruikt, zoals de kolommethode of de nucleïnezuurprecipitatiemethode, is de kans groot dat de weinige monsters verloren gaan.Een oplossing is om een dragermolecuul toe te voegen, zoals tRNA, maar zelfs dan is er geen garantie dat ons herstelexperiment in orde is.
Dus wat is een betere manier?Een goede optie voor gekweekte cellen of microanatomische monsters is het gebruik van directe lysis.
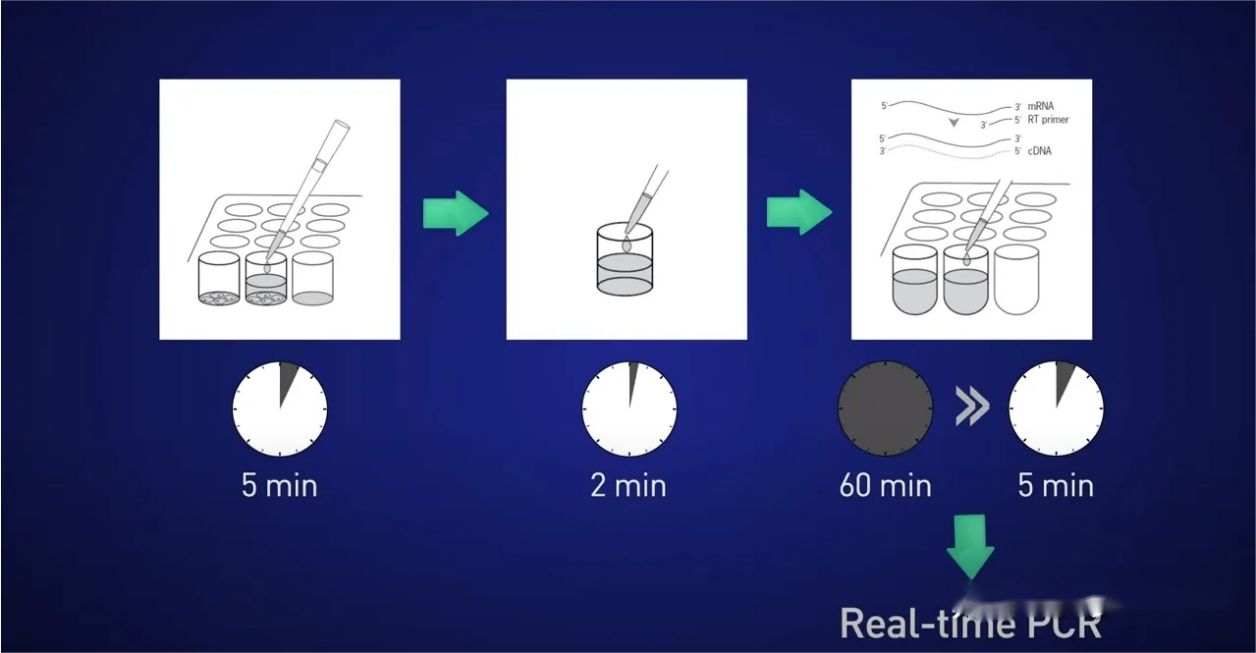
Het idee is om de cellen gedurende 5 minuten te splitsen, het RNA in de oplossing los te laten, dan de reactie gedurende 2 minuten te stoppen, dan het lysaat direct toe te voegen aan de omgekeerde transcriptiereactie zodat er geen RNA verloren gaat, en tenslotte het resulterende cDNA rechtstreeks in de oplossing te plaatsen. in de real-time reactie.
Maar wat als we, vanwege een beperkt startpunt of een kleine hoeveelheid doelwitgenexpressie, al het RNA kunnen recyclen en toch niet genoeg sjablonen kunnen leveren om een goed real-time signaal te krijgen?
In dit geval kan de voorversterkingsstap erg nuttig zijn.
Het volgende is een schema om de gevoeligheid na omgekeerde transcriptie te verhogen.Voordat we beginnen, moeten we stroomafwaarts vragen in welke doelen we geïnteresseerd zijn, om specifieke primers voor deze doelen te ontwerpen voor pre-amplificatie.
Dit kan worden bereikt door een gemengde primer te maken met maximaal 100 paar primers en een reactiecyclus van 10 tot 14 keer.Daarom is een speciaal voor deze eis ontworpen mastermix nodig om het verkregen cDNA voor te amplificeren.
De reden voor het instellen van het aantal cycli tussen 10 en 14 is dat dit beperkte aantal cycli zorgt voor willekeur tussen de verschillende targets, wat cruciaal is voor onderzoekers die kwantitatieve moleculaire informatie nodig hebben.
Na pre-amplificatie kunnen we een grote hoeveelheid cDNA krijgen, zodat de detectiegevoeligheid aan de achterkant sterk wordt verbeterd, en we kunnen zelfs het monster verdunnen en meerdere real-time PCR-reacties uitvoeren om mogelijke willekeurige fouten te elimineren.
Posttijd: 11 april 2023